Post-processing for 3D feature Map
我使用的 KL520 的dongle
想要了解 3d feature map是是怎透過post processing產生的
並比較 python上的output 與 KL520 dongle的output 浮點數的結果
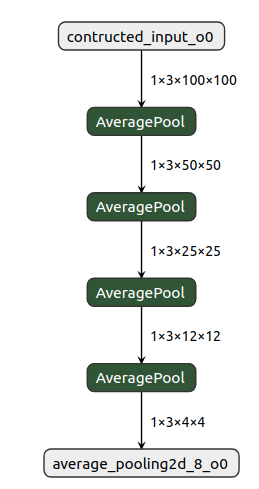
我用keras建構以下的簡單model,output feature map 為 1*3*4*4 (channel first)
我使用11張自己設定的 100*100的jpg圖來compile NEF檔案,再faAnalyserCompiler過程中,遇到以下warning
不過似乎沒有太大問題,最後有產生nef檔案。
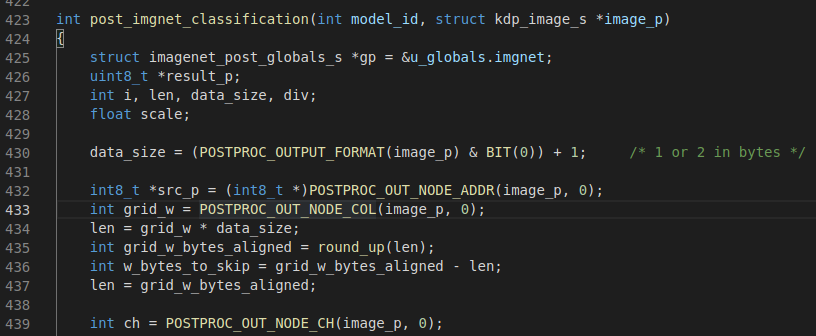
我觀察 example/post_processing_ex.c 中的 post_imgnet_classification函數
知道post_processing是透過POSTPROC_OUT_NODE_ADDR() 來得到 image_p 的指標 src_p
然後以下就看不太懂了,433~437 行似乎是在做一些對齊的動作,計算shift,接著再用src_p來取得浮點數的資資料
不知道我理解是否有錯?
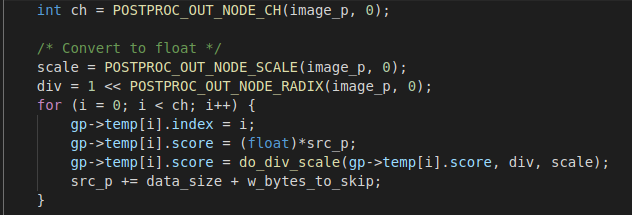
然後我不太懂以下程式碼,尤其是w_bytes_to_skip,為何這樣shift就能取channel的資料呢??
最主要的問題就是 src_p 是如何存放feature map 的float資料的,(C/H/W)是怎樣放的
我要怎樣才可以把src_p這個很麻煩的指標,轉成比較好理解的3d array 每個element都是float,
這樣可以方便與python 比較結果,能確定model轉換後沒錯誤,也可以比較float feature map 的差異,來判斷model compile 後是否有太多的精度損失
Tagged:
The discussion has been closed due to inactivity. To continue with the topic, please feel free to post a new discussion.


Comments
Hello,
在KL520上,若是使用DME的API來做推論,所拿到的結果格式將會如下,可參考章節7.2中Post Process的介紹http://doc.kneron.com/docs/#520_1.5.0.0/getting_start/#7-run-model-binaries-in-dme-mode
total_out_number + (c/h/w/radix/scale) + (c/h/w/radix/scale) + ... + fp_data + fp_data + ...
其中,total_out_number表示你的模型最終會有幾組output,在你的模型中只會有一組 (average_pooling2d_8_o0),接著依照output的數量,每組output會有一組資訊來描述該組output的channel, height, weight, radix, scale,你說看到的POSTPROC_OUT_XXX()其實都是在dme拿到結果後先將值填入,方便後面透過marco取值,填入的部分可以參考範例 kl520_isi_async_bypass_pre_post_host_yolo.cpp
後面才是接著第一組第一組feature map的資料,但會以定點(fixed point)的方式表示,定點轉換浮點的公式如下
float_value = do_div_scale_2( (float) fixed_value, fScale )
fScale的計算方式如下,需透過前述的radix與scale來計算
div = 1 << radix
fScale = 1 / (div * scale);
函式內容可參考 postprocess_ex.c中的do_div_scale_2()
另外,因為硬體架構的緣故,在取值的時候有兩個部分要特別注意
也就是說,即便你的output c, w, h是3x4x4,在KL520上輸出的結果會變成 c, h, w = 3x4x16,這部分要自己重新排列成想要的格式
非常感謝您的回覆,我已經搞清楚src_p的格式了
我用以下的程式片段來將解析 3d feature map,再這裡分享給其他需要的人
float 解析完之後,存在 res_float_array 這個 float *
感謝分享!
在接下來新版SDK的release中,我們也會提供相關格式的輸出直接供大家使用